For litt over en måned siden, rapporterte vi om fremskritt i å bruke DNA som et høy-kapasitets informasjons-lagringsmedium , og spådde at "racet om bedre DNA- lagringsprodukter vil akselerere når det beveger seg fra laboratoriet til markedet." I dag er det flere nyheter i det markedskreftene tråkker på gassen. Enda mer spennende, finner DNA bruksområder for mer enn bare lagring av data.
DNA i informasjonsteknologi
En stor forbedring i DNA informasjons lagring og gjenfinning har blitt annonsert i Science Magazine. To forskere fra Columbia Universitet og New York Genome Center rapporterte et nytt høyt resultat for DNA datalagring - over to størrelsesordener bedre enn tidligere forsøk. De var i stand til å kode tekst, bilder, film, og et operativsystem i 2 megabyte med DNA, og hente den tilbake helt i flere studier. Nyheter fra 'Columbia University data Science Institute' sier her:
"Menneskeheten kan fort generere mer data enn harddisker eller magnetbånd kan håndtere, et problem som får forskerne til å vende seg til naturens eldgamle løsning for informasjonslagring - DNA".
I en ny studie i Science, et par forskere ved Columbia University og New York Genome Senter (NYGC) viser at en algoritme konstruert for å streame video på en mobiltelefon kan låse opp DNA's nesten fulle lagringspotensiale ved å klemme mer informasjon inn i dens fire basis nukleotider. De viser at denne teknologien er også svært pålitelig.
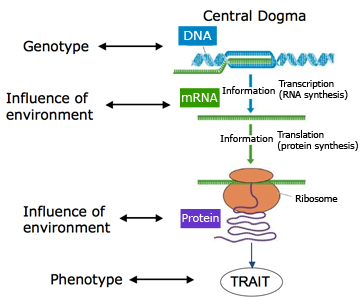 I et videoklipp i artikkelen beskriver Yaniv Ehrlich hva de gjorde og hvorfor det er viktig. Videoen har moro med ordene: “Alle dine data i en dråpe av DNA." Informasjonen lagringskapasitet av DNA, dets kompakthet og holdbarhet gjør det svært ønskelig for den neste generasjonen av datamaskiner. Det kan vare i flere tusen år hvis de oppbevares på et kjølig og tørt sted.
I et videoklipp i artikkelen beskriver Yaniv Ehrlich hva de gjorde og hvorfor det er viktig. Videoen har moro med ordene: “Alle dine data i en dråpe av DNA." Informasjonen lagringskapasitet av DNA, dets kompakthet og holdbarhet gjør det svært ønskelig for den neste generasjonen av datamaskiner. Det kan vare i flere tusen år hvis de oppbevares på et kjølig og tørt sted.
"DNA vil ikke brytes ned over tid som kassetter og CD-er, og det vil ikke bli foreldet - hvis det gjør, har vi større problemer," sier studie-medforfatter Yaniv Ehrlich, en professor i datavitenskap ved Columbia Engineering, et medlem av Columbias data Science Institute, og et fast medlem av NYGC.
Ehrlich og Zielinsky beskriver hvorfor den praktiske grensen for Shannon informasjon i DNA er 1,8 bits per nukleotid (ikke to som forventet,) på grunn av indekserings og feilkorreksjons krav. Algoritmen deres oppnådde 1,6 bits pr nukleotide, ganske nær den teoretiske grensen. De komprimerte først alle sine innspill i et enkelt Linux arkiv og hakket det i 32-tegns segmenter. For å oversette binære tall til DNA, skapte de en enkel regel som representerer par av binære tall (00, 01, 10, 11) som de fire DNA-nukleotider (A, C, G, T). Etter oversettelsen, sendte de kodene over hele USA til et selskap for å generere DNA. I alt genererte de en digital liste bestående av 72.000 DNA-tråder, hver 200 baser lang, og sendte det i en tekstfil til en San Francisco DNA-syntese oppstart, Twist Bioscience, som spesialiserer seg på å omforme digitale data til biologiske data. To uker senere fikk de en ampulle som inneholdt en spesifikasjon av DNA-molekyler.
Bilde 2. DNA -samspill med omgivelsene
For å hente filene sine, brukte de moderne sekvenseringsteknologi til å lese DNA-trådene, etterfulgt av programvare for å oversette den genetiske koden tilbake til binær. De gjenopprettet filene sine med null feil, melder studien. (I denne korte demoen , åpner Ehrlich sitt arkivert operativsystem på en virtuell maskin og spiller et spill Minesveiper for å feire.)
Deres suksess er avhengig av en teknikk som kalles "kildekode" (se artikkel fra Harvard) som er mye brukt av streaming teknologier. I motsetning til tidligere overførings algoritmer, trenger kildekodene ikke å kreve at korrupte eller manglende pakker skal sendes på nytt. Algoritmen bare sprayer ut data-pakkene som dråper i en fontene. Ehrlich kaller selv pakkene i sine "DNA Fontene" dråper, som består av DNA-oligo-nukleotider med fast lengde (200 baser). Vi bør merke oss at disse dråpene ble angitt i "DNA tekst" før de ble omgjort til faktiske molekyler.
 I følge med hver dråpe er en 4-bits "frø" eller strekkode med ekstra informasjon, levert av en overførings-algoritme, først brukt til inndata, pluss to biter for feilretting. De ekstra bitene gjør det mulig for mottakeren å rekonstruere sekvensen når den blir hentet. Denne lille mengden av merinformasjon sikrer feilfri rekonstruksjon av den opprinnelige informasjonen - det være seg bilder, filmer, musikk, tekst, eller endog et operativsystem.
I følge med hver dråpe er en 4-bits "frø" eller strekkode med ekstra informasjon, levert av en overførings-algoritme, først brukt til inndata, pluss to biter for feilretting. De ekstra bitene gjør det mulig for mottakeren å rekonstruere sekvensen når den blir hentet. Denne lille mengden av merinformasjon sikrer feilfri rekonstruksjon av den opprinnelige informasjonen - det være seg bilder, filmer, musikk, tekst, eller endog et operativsystem.
Bilde 3. Binær DNA-koding
I tillegg til høyere lagringstetthet, nådde Columbia teamet også nye høyder med pålitelighet. De demonstrerte metodens robusthet ved å lage kopier av kopier sju ganger i serie. Selv etter denne "dyp-kopieringen," var de i stand til å hente ut dataene uten feil.
 For å oppsummere, i dette arbeidet, rapporterte vi en effektiv og robust koding strategi som tillater nesten ubegrenset data gjenoppretting og høy fysisk tetthet mens vi nærmer oss Shannon kapasiteten for DNA lagring, nærmere enn for noen tidligere design. Vi testet vårt rammeverk med en forholdsvis stor fil sammenlignet med de som ble brukt i tidligere studier, og var i stand til helt å gjenopprette data under forskjellige tester. I det vi implementerte vår tilnærming i symfoni med langsiktige bevarings teknikker, som for eksempel DNA innebygging i silika perler, noe som kan kreve ytterligere finjustering av redundans nivåer. Vi forventer at en slik finjustering kan dra nytte av høy fleksibilitet i DNA- Kilde-rammeverket, som tillater bestemmelse av praktisk talt alle redundans nivåer uten å endre programvare eller påvirke dekodingstiden.
For å oppsummere, i dette arbeidet, rapporterte vi en effektiv og robust koding strategi som tillater nesten ubegrenset data gjenoppretting og høy fysisk tetthet mens vi nærmer oss Shannon kapasiteten for DNA lagring, nærmere enn for noen tidligere design. Vi testet vårt rammeverk med en forholdsvis stor fil sammenlignet med de som ble brukt i tidligere studier, og var i stand til helt å gjenopprette data under forskjellige tester. I det vi implementerte vår tilnærming i symfoni med langsiktige bevarings teknikker, som for eksempel DNA innebygging i silika perler, noe som kan kreve ytterligere finjustering av redundans nivåer. Vi forventer at en slik finjustering kan dra nytte av høy fleksibilitet i DNA- Kilde-rammeverket, som tillater bestemmelse av praktisk talt alle redundans nivåer uten å endre programvare eller påvirke dekodingstiden.
Bilde 4. Ulike nivåer av koding i DNA
Vil bedrifter flokke seg om denne teknologien? De viktigste begrensninger er kostnad og hastighet. Men husk du de første silisium datamaskinene? Fordi DNA er så attraktiv, kan vi sikkert forvente at oppfinnere finner raskere, billigere måter å syntetisere vanlige DNA-tråder. Lesning ut av DNA med PCR og nyskapende sekvens-teknikker har allerede blitt billig og enkelt siden 'Human Genome Project' fikk det til å virke som en formidabel "månelandings" prosjekttype. Forfatterne forventer at entreprenører vil finne "optimaliseringspunktet som kombinerer den høyeste kjemiske gjennomstrømning med høyest mulig hastighet (biter per nukleotid) av designkoding." Når det skjer, "kan DNA bli en økonomisk levedyktig løsning for langsiktig høy kvalitetslagring. "
Mens vi vurderer lagring, hvorfor ikke vurdere databehandling med DNA? En kort artikkel om Futurism.com annonserer, "selvreproduserende 'DNA Computers' er satt til å forandre alt -her." Les om hvordan forskere ved University of Manchester "arbeider med å få tråder av DNA inn i neste grunnlag for databehandling." Fordi DNAs doble heliks kan ta to baner samtidig, så kan det løse problemer raskere. Det er artig, men tenk om fremgang i biologiske inndata og utdata teknolog en gang kan tillate DNA-datamaskiner å få tilgang til DNA lagring. (Hei, er det hva som skjer i livet?)
 DNA i elektroteknikk
DNA i elektroteknikk
Visste du at DNA leder strøm? Nyheter fra Arizona State begynner med dette bemerkelsesverdig uttalelse: "DNA, livets stoff, kan meget vel også ordne problemet for ingeniører som prøver å fremme utviklingen av små og lave kostnader for elektroniske enheter." Et team ledet av Nongjiang Tao lyktes i å lage en styrbar elektrisk bryter ut av DNA som er 1000 ganger tynnere enn et menneskelig hårstrå. Du kan lese om deres "ingeniørbragd" i open-access tidsskrift Nature Communications.
Bilde 5. Elektronisk krets
Se også "Switched-on-DNA" i en artikkel fra ASU's velrenomerte Biomimetics divisjon, Biodesign Institute . Denne multi-million-dollar instituttet, med 800.000 kvadratfot med forsknings plass og over 600 ansatte og 62 faste professorer (én som er Nobelprisvinner), er et godt eksempel på 'gullrushet' i "bioinspirert innovasjon." Se etter intelligent design begreper i oversikts-siden (under):
"Laget på premisset om at forskere kan overvinne komplekse samfunns-problemer ved å gjenskape "design regler" som finnes i naturen, adresserer instituttets forskere et ekspansivt område av globale utfordringer ved å skape "bioinspirerte løsninger, "....
[Mål-erklæring:] Biodesign Institutet ved ASU adresserer dagens kritiske globale utfordringer i helsevesenet, bærekraft og sikkerhet ved å utvikle løsninger som er inspirert av naturlige systemer og oversette disse løsningene til kommersielt levedyktige produkter og klinisk praksis.
DNA i mekanisk ingeniørkunst
Til slutt, forrige måned nevnte vi at et team fra Purdue Universitetet, "inspirert av naturlige biologiske motorer" som kinesin, laget en egen "DNA-spaserer". De ser for seg å bruke slike syntetiske molekylære maskiner i biomedisinske og industrielle applikasjoner, kanskje til og med som kjøretøy for å levere ut medisiner mot kreft inne i kroppen.
 Et bemerkelsesverdig molekyl
Et bemerkelsesverdig molekyl
Bilde 6. DNA -livets molekyl
Den lagrer informasjon, det leder strøm, og det beveger seg. Det faktum at DNA's sekvenser av molekylære baser kan representeres med bokstaver (A, C, T, G), konvertert til binære siffer (0, 1), og leses i en datamaskin som kan starte den opp som et operativsystem og spille en film er virkelig spennende. Ehrlich og Zielinski tilstand at den teoretiske informasjonslagringskapasiteten på DNA er 680 petabyte (680 x 10 opphøyd i 15) per gram. Forestill 680 millioner milliarder tegn med informasjon i en gjenstand med vekt til en binders! Ingen flere lange korridorer med harddisker; verdens informasjon vil passe i ett rom, eller til og med en skoeske.
Teoretisk sett, ser vi som en sideeffekt, kan enkeltatomer være bærere av informasjon. Men en gjennomgang i Nature så på en fersk test av dette, og besluttet at det bare kunne skje under ekstreme forhold ", slik som i en ultrahøyt vakuum.” Atomet måtte være montert på en molekylær overflate uansett. I tillegg vil alvorlige begrensninger på atomnivå måtte forseres for pålitelig å kode og dekode informasjon: kontrollere magnetisk isotropi, molekylære vibrasjoner, og interaksjoner mellom molekylet og dets omgivelser. "Dette er kvantitative krav hvis oppfyllelse ikke vil være rett fram", så ikke se for deg fremgang av den skala i rimelig framtid.
 Tilbake til DNA- nøkkelen til å forstå nytteverdien er dens lagrede sekvens. Tilfeldige nukleotider gjør ingenting. De formidler ikke informasjon, de går ikke , og de danner ikke elektriske kretser. Bare ved intelligent designede algoritmer for nøye å kontrollere koding og dekoding av sekvensen, var disse ingeniørene i stand til å fullføre det de gjorde.
Tilbake til DNA- nøkkelen til å forstå nytteverdien er dens lagrede sekvens. Tilfeldige nukleotider gjør ingenting. De formidler ikke informasjon, de går ikke , og de danner ikke elektriske kretser. Bare ved intelligent designede algoritmer for nøye å kontrollere koding og dekoding av sekvensen, var disse ingeniørene i stand til å fullføre det de gjorde.
Ehrlich og Zielinsky snakket om “evolusjonært optimalisert maskineri" som trofast replikerer informasjon. Kanskje måtte de si det for å passere AAAS sin fagfellevurdering (?) Vi forventer imidlertid at de ikke ville være fornøyd om noen vendte det mot dem, og sa at deres prestasjon ble gjort av blinde, ikke-styrte prosesser. Nei; DNA er, på en måte, bedre forstått som "Designede Naturlige Algoritmer."
Bilde 7. Programmering av Designet Naturlig Alogritme
Oversatt av Asbjørn E. Lund
(Bildene sto ikke i opprinnelig artikkel, evt. untatt det 1., og er satt inn av undertegnede, se lenke i Bilde-nr)
 Den hotteste nye datamaskinen: DNA
Den hotteste nye datamaskinen: DNA Den hotteste nye datamaskinen: DNA
Den hotteste nye datamaskinen: DNA I et videoklipp i artikkelen beskriver Yaniv Ehrlich hva de gjorde og hvorfor det er viktig. Videoen har moro med ordene: “Alle dine data i en dråpe av DNA." Informasjonen lagringskapasitet av DNA, dets kompakthet og holdbarhet gjør det svært ønskelig for den neste generasjonen av datamaskiner. Det kan vare i flere tusen år hvis de oppbevares på et kjølig og tørt sted.
I et videoklipp i artikkelen beskriver Yaniv Ehrlich hva de gjorde og hvorfor det er viktig. Videoen har moro med ordene: “Alle dine data i en dråpe av DNA." Informasjonen lagringskapasitet av DNA, dets kompakthet og holdbarhet gjør det svært ønskelig for den neste generasjonen av datamaskiner. Det kan vare i flere tusen år hvis de oppbevares på et kjølig og tørt sted. I følge med hver dråpe er en 4-bits "frø" eller strekkode med ekstra informasjon, levert av en overførings-algoritme, først brukt til inndata, pluss to biter for feilretting. De ekstra bitene gjør det mulig for mottakeren å rekonstruere sekvensen når den blir hentet. Denne lille mengden av merinformasjon sikrer feilfri rekonstruksjon av den opprinnelige informasjonen - det være seg bilder, filmer, musikk, tekst, eller endog et operativsystem.
I følge med hver dråpe er en 4-bits "frø" eller strekkode med ekstra informasjon, levert av en overførings-algoritme, først brukt til inndata, pluss to biter for feilretting. De ekstra bitene gjør det mulig for mottakeren å rekonstruere sekvensen når den blir hentet. Denne lille mengden av merinformasjon sikrer feilfri rekonstruksjon av den opprinnelige informasjonen - det være seg bilder, filmer, musikk, tekst, eller endog et operativsystem. For å oppsummere, i dette arbeidet, rapporterte vi en effektiv og robust koding strategi som tillater nesten ubegrenset data gjenoppretting og høy fysisk tetthet mens vi nærmer oss Shannon kapasiteten for DNA lagring, nærmere enn for noen tidligere design. Vi testet vårt rammeverk med en forholdsvis stor fil sammenlignet med de som ble brukt i tidligere studier, og var i stand til helt å gjenopprette data under forskjellige tester. I det vi implementerte vår tilnærming i symfoni med langsiktige bevarings teknikker, som for eksempel DNA innebygging i silika perler, noe som kan kreve ytterligere finjustering av redundans nivåer. Vi forventer at en slik finjustering kan dra nytte av høy fleksibilitet i DNA- Kilde-rammeverket, som tillater bestemmelse av praktisk talt alle redundans nivåer uten å endre programvare eller påvirke dekodingstiden.
For å oppsummere, i dette arbeidet, rapporterte vi en effektiv og robust koding strategi som tillater nesten ubegrenset data gjenoppretting og høy fysisk tetthet mens vi nærmer oss Shannon kapasiteten for DNA lagring, nærmere enn for noen tidligere design. Vi testet vårt rammeverk med en forholdsvis stor fil sammenlignet med de som ble brukt i tidligere studier, og var i stand til helt å gjenopprette data under forskjellige tester. I det vi implementerte vår tilnærming i symfoni med langsiktige bevarings teknikker, som for eksempel DNA innebygging i silika perler, noe som kan kreve ytterligere finjustering av redundans nivåer. Vi forventer at en slik finjustering kan dra nytte av høy fleksibilitet i DNA- Kilde-rammeverket, som tillater bestemmelse av praktisk talt alle redundans nivåer uten å endre programvare eller påvirke dekodingstiden. DNA i elektroteknikk
DNA i elektroteknikk Et bemerkelsesverdig molekyl
Et bemerkelsesverdig molekyl Tilbake til DNA- nøkkelen til å forstå nytteverdien er dens lagrede sekvens. Tilfeldige nukleotider gjør ingenting. De formidler ikke informasjon, de går ikke , og de danner ikke elektriske kretser. Bare ved intelligent designede algoritmer for nøye å kontrollere koding og dekoding av sekvensen, var disse ingeniørene i stand til å fullføre det de gjorde.
Tilbake til DNA- nøkkelen til å forstå nytteverdien er dens lagrede sekvens. Tilfeldige nukleotider gjør ingenting. De formidler ikke informasjon, de går ikke , og de danner ikke elektriske kretser. Bare ved intelligent designede algoritmer for nøye å kontrollere koding og dekoding av sekvensen, var disse ingeniørene i stand til å fullføre det de gjorde.